GenomeLab™ GeXPのプライマー伸長による1反応で最大10SNP検出、または未知のアレル検出
SNPにはいくつかの選択肢があります。
一塩基多型(SNP)は遺伝子変異の主要な役割を占めており、既知の遺伝子多型の約80%を構成しています。ヒトゲノムにおける量は平均で1000塩基対あたり1と推定されています。SNPのほとんどは二対立遺伝子(Short Tandem Repeats(STR)よりも情報量が少ない)ですが、頻度が高く変異的には安定しており、マーカーと未知の変異間の連鎖不平衡(LD)が疾患を引き起こす突然変異のマッピングに使用されている関連研究に適しています。
GenomeLab GeXP遺伝子解析システムはSNP解析に2つの方法を提供します。Single-Base Extension(一塩基伸長法)解析とDNAシークエンシングGenomeLabソフトウェアは、シングルもしくはマルチプレックスのSNP産物に対し、自動サイジングとアレルコーリングを提供します。ソフトウェアはSTR遺伝子座と同様のメカニズムを使用してSNP遺伝子座を解析します。
プライマー伸長によるSNPスコアリング
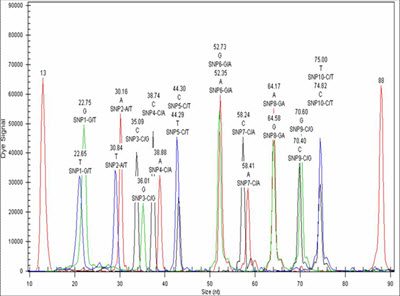
GeXPシステムは1つのテンプレート(または異なる複数のテンプレート)の複数のサイトでSNPの同時解析を実行可能にします。 GenomeLab™ SNPStart Primer Extension Kitは一塩基プライマー伸長法(SNP)に基づく業界標準です。1回の反応で最大10SNPのマルチプレックスが検証、最適化されたSNPStart Kitは低から中程度のスループットアプリケーションにとって理想的です。SNP遺伝子型は自動SNPローカスタグ割り当てを通じてGeXPシステムのフラグメント解析ソフトウェアによりレポートが作成されます。
GenomeLab SNPStart Primer Extensionキットを使用したマルチプレックスSNPスコアリング解析
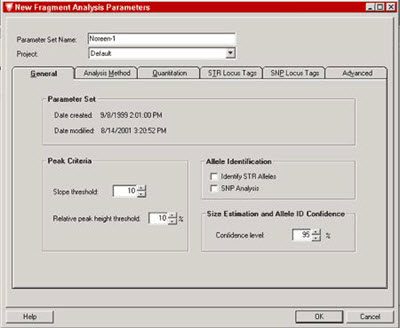
結果に関連するすべてのパラメーターを指先一つで操作し、データを簡単にレビューできるようにします。エレクトロフェログラムはスタックやオーバーレイ表示によりデータの比較をしやすくできます。
アレルはサイズまたはサイズと色に基づいて同定され、SNPは同じ簡単な設定で同定できます。
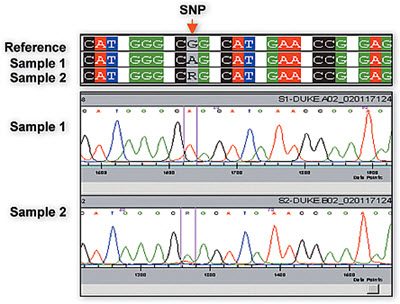
シークエンシングによるSNPの発見
DNAシークエンシングは未知のアレルを発見する主要な方法です。GeXP遺伝子解析システムはダイターミネーターサイクルシークエンシングを使用する効率的で堅牢なDNAシークエンシングプロトコルを提供します。未知のサンプルのシークエンスはリファレンス配列とアライメントされ、SNPの存在するローカスが同定されます。